На этом шаге мы рассмотрим реализацию такого сжатия на конкретном примере.
Зачастую важна бывает экономия места - виртуального или реального. Использовать меньше места означает более эффективно работать и экономить деньги. Если вы арендуете квартиру большей площади, чем нужно для ваших семьи и вещей, то можете ужаться до жилья меньшего размера, которое стоит дешевле. Если вы побайтно платите за хранение данных на сервере, то можете сжать данные так, чтобы их хранение обходилось дешевле. Сжатие - это процесс получения данных и их кодирования (изменения формы) таким образом, чтобы они занимали меньше места. Распаковка предусматривает обратный процесс - возвращение данных в исходную форму.
Если сжатие данных так эффективно для их хранения, то почему оно не применяется для всех данных? Дело в том, что существует компромисс между временем и пространством. На то, чтобы сжать часть данных и распаковать их обратно в исходную форму, требуется время. Поэтому сжатие данных имеет смысл только в ситуациях, когда небольшой размер важнее, чем быстрое выполнение. Возьмем, к примеру, большие файлы, передаваемые через Интернет. Их сжатие имеет смысл, поскольку для передачи файлов потребуется больше времени, чем для их распаковки после получения. Кроме того, время, необходимое для сжатия файлов при их хранении на исходном сервере, необходимо учитывать только один раз.
Сжать данные самыми простыми способами можно тогда, когда вы понимаете, что типы хранилищ данных используют больше битов, чем необходимо для их содержимого. Например, на низком уровне, если целые числа без знака, значения которых никогда не превысят 65535, сохраняются в памяти как 64-разрядные целые числа без знака, то они сохраняются неэффективно. Вместо этого их можно хранить как 16-разрядные целые числа без знака. Это уменьшит потребление пространства для фактического хранения чисел на 75% (16 бит вместо 64). Таким неэффективным способом хранятся миллионы чисел, это может означать до мегабайта впустую потраченного пространства.
В Python из соображений простоты (что, конечно же, является законной целью) разработчик иногда избавлен от размышлений. Не существует типов 64-разрядных целых чисел без знака и 16-разрядных целых чисел без знака. Есть только один тип int, который позволяет хранить числа произвольной точности. Узнать, сколько байтов памяти потребляют ваши объекты Python, поможет функция sys.getsizeof(). Но из-за вычислительных издержек, свойственных объектной системе Python, в Python 3.7 нет способа создать тип int, который занимал бы менее 28 байт (224 бита). Для хранения одного числа типа int можно добавлять по одному биту за раз (как мы и сделаем в этом примере), но оно все равно занимает как минимум 28 байтов.
 Если вы давно не имели дела с двоичными числами, напомним, что бит - это одно значение, равное 1 или 0. Последовательность нулей и единиц позволяет представить число в системе счисления по
основанию 2. Для целей этого раздела вам не нужно выполнять какие-либо математические операции в двоичной системе счисления, но вы должны понимать, что число битов, которые способен
хранить тип, определяет то, сколько разных значений в нем можно представлять. Например, 1 бит может представлять два значения (0 или 1), 2 бита могут представлять четыре значения (00, 01,
10, 11), 3 бита - восемь значений и т. д.
Если вы давно не имели дела с двоичными числами, напомним, что бит - это одно значение, равное 1 или 0. Последовательность нулей и единиц позволяет представить число в системе счисления по
основанию 2. Для целей этого раздела вам не нужно выполнять какие-либо математические операции в двоичной системе счисления, но вы должны понимать, что число битов, которые способен
хранить тип, определяет то, сколько разных значений в нем можно представлять. Например, 1 бит может представлять два значения (0 или 1), 2 бита могут представлять четыре значения (00, 01,
10, 11), 3 бита - восемь значений и т. д.
Если число различных значений, которые способен представлять тип, меньше, чем число значений, которые могут представлять биты, используемые для его хранения, то такой тип может быть сохранен более эффективно. Рассмотрим, к примеру, нуклеотиды, которые образуют ген в ДНК (Этот пример взят из книги: Седжвик Р., Уэйн К. Алгоритмы на Java. 4-е изд. - М.: Вильямс, 2013 (Sedgewick R., Wayne К. Algorithms. 4th ed. - Addison-Wesley Professional, 2011)). Каждый нуклеотид может принимать только одно из четырех значений: А, С, G или Т. (Подробнее об этом будет рассказано в следующих шагах.) Однако, если ген хранится как тип str, что можно рассматривать как коллекцию символов Unicode, каждый нуклеотид будет представлен символом, для хранения которого обычно требуется 8 бит. В двоичном коде для хранения типа с четырьмя возможными значениями требуется всего 2 бита. Значения 00, 01, 10 и 11 - это четыре различных значения, которые могут быть представлены 2 битами. Если А соответствует 00, С - 01, G - 10, а Т - 11, то объем хранилища, необходимого для строки нуклеотидов, может быть уменьшен на 75% (с 8 до 2 бит на каждый нуклеотид).
Вместо того чтобы хранить нуклеотиды как тип str, их можно сохранить как строку битов (рисунок 1).
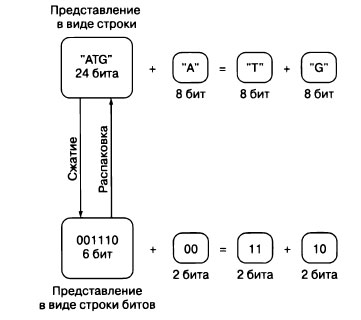
Рис.1. Сжатие типа str, представляющего ген, в строку битов, где выделяется по 2 бита на нуклеотид
Строка битов - это именно то, чем она кажется: последовательность нулей и единиц произвольной длины. К сожалению, стандартная библиотека Python не содержит готовых конструкций для работы с битовыми строками произвольной длины.
Следующий код преобразует строку, состоящую из букв А, С, G и Т, в строку битов и обратно. Строка битов хранится в виде числа int. Поскольку тип int в Python может быть произвольной длины, его можно использовать как строку битов произвольной длины. Чтобы преобразовать ее обратно в str, реализуем на Python специальный метод __str__().
class CompressedGene: def __init__(self, gene: str) -> None: self._compress(gene)
В классе CompressedGene предоставляется строка символов типа str, описывающих нуклеотиды в гене. Внутри класса хранится последовательность нуклеотидов в виде строки битов. Основное назначение метода __init__() состоит в инициализации конструкции строки битов соответствующими данными. Метод __init__() вызывает _compress(), который выполняет всю грязную работу по фактическому преобразованию предоставленной строки нуклеотидов str в строку битов.
Обратите внимание на то, что имя _compress() начинается с подчеркивания. В Python нет концепции настоящих закрытых методов или переменных. (Все переменные и методы могут быть доступны через рефлексию, строгого соблюдения приватности здесь нет.) По соглашению знак подчеркивания в начале имени указывает на то, что акторы за пределами класса не должны полагаться на реализацию этого метода. (Впоследствии это может быть изменено, и такие методы должны рассматриваться как приватные.)
 Если имя метода или переменной в классе начинается с двух символов подчеркивания, то Python будет искажать это имя, изменяя его имя реализации с помощью "соли", что не позволит
другим классам легко его обнаруживать. Здесь используется одинарное подчеркивание для обозначения приватных переменных и методов, но вы можете применять двойное, если действительно хотите подчеркнуть,
что элемент является приватным. Подробнее об именовании в Python читайте в разделе "Описательные стили именования" в РЕР 8: https://peps.python.org/pep-0008/#descriptive-naming-styles.
Если имя метода или переменной в классе начинается с двух символов подчеркивания, то Python будет искажать это имя, изменяя его имя реализации с помощью "соли", что не позволит
другим классам легко его обнаруживать. Здесь используется одинарное подчеркивание для обозначения приватных переменных и методов, но вы можете применять двойное, если действительно хотите подчеркнуть,
что элемент является приватным. Подробнее об именовании в Python читайте в разделе "Описательные стили именования" в РЕР 8: https://peps.python.org/pep-0008/#descriptive-naming-styles.
Теперь посмотрим, как на самом деле можно выполнить сжатие.
def _compress(self, gene: str) -> None:
self.bit_string: int = 1 # начальная метка
for nucleotide in gene.upper():
self.bit_string <<= 2 # сдвиг влево на 2 бита
if nucleotide == "A": # поменять 2 последних бита на 00
self.bit_string |= 0b00
elif nucleotide == "C": # поменять 2 последних бита на 01
self.bit_string |= 0b01
elif nucleotide == "G": # поменять 2 последних бита на 10
self.bit_string |= 0b10
elif nucleotide == "T": # поменять 2 последних бита на 11
self.bit_string |= 0b11
else:
raise ValueError("Плохой нулкеотид: {}".format(nucleotide))
Метод _compress() последовательно просматривает каждый символ в строке нуклеотидов типа str. Встретив символ А, он добавляет в строку битов 00, встретив С - 01 и т. д. Помните, что каждый нуклеотид занимает 2 бита. Поэтому перед добавлением очередного нуклеотида мы сдвигаем битовую строку на 2 бита влево (self.bit_string <<= 2).
Каждый нуклеотид добавляется с помощью операции ИЛИ (|). После сдвига влево с правой стороны строки битов добавляются два нуля. Если в побитовых операциях ИЛИ, таких как self.bit_string |= 0b10, операндами являются ноль и любое другое значение, то это значение заменяет 0. Другими словами, мы постоянно добавляем 2 новых бита в правую часть строки битов. Два добавляемых бита определяются типом нуклеотида.
Наконец, мы реализуем декомпрессию и специальный метод __str__(), который ее использует.
def decompress(self) -> str:
gene: str = ""
for i in range(0, self.bit_string.bit_length() - 1, 2):
# -1 чтобы исключить метку
bits: int = self.bit_string >> i & 0b11
# получить только 2 значимых бита
if bits == 0b00: # А
gene += "A"
elif bits == 0b01: # C
gene += "C"
elif bits == 0b10: # G
gene += "G"
elif bits == 0b11: # T
gene += "T"
else:
raise ValueError("Плохой бит:{}".format(bits))
return gene[::-1] # [::-1] обращение строки посредством обратных срезов
def __str__(self) -> str: # представление строки в биде красивого вывода
return self.decompress()
Функция decompress() считывает из строки битов по 2 бита и использует их, чтобы определить, какой символ добавить в конец представления гена типа str. Поскольку биты считываются в обратном направлении, по сравнению с последовательностью, в которой они были сжаты (справа налево, а не слева направо), то представление str в итоге обращается (с помощью нотации срезов для обращения [::-1]). Наконец, обратите внимание на то, как удобный метод bit_length() типа int помог нам при разработке decompress(). Проверим, как это работает.
if __name__ == "__main__": from sys import getsizeof original: str = "TAGGGATTAACCGTTATATATATATAGCCATGGATCGATTATATAGGGATTAACCGT TATATATATATAGCCATGGATCGATTATA" * 100 print("Исходный: {} бит".format(getsizeof(original))) compressed: CompressedGene = CompressedGene(original) # сжатие print("Упакованный: {} бит".format(getsizeof(compressed.bit_string))) print(compressed) # распаковка print("Исходный и упакованный одинаковые: {}".format(original == compressed.decompress()))

Рис.2. Результат работы приложения
Используя метод sys.getsizeof(), можно указать при выводе, действительно ли мы сэкономили почти 75% места в памяти при сохранении гена с помощью этой схемы сжатия.
 В классе CompressedGene мы широко задействовали операторы if для выбора разных вариантов методов сжатия и распаковки. Это довольно типично для Python, поскольку
в нем нет оператора switch. Вы также увидите, что для выбора из множества вариантов Python часто основательно опирается на словари вместо интенсивного использования операторов if.
Представьте себе, например, словарь, в котором мы могли бы находить соответствующую комбинацию битов для каждого нуклеотида. Иногда это выглядит более читабельным, но может снизить
производительность. Несмотря на то что с технической точки зрения сложность поиска по словарю равна O(1), затраты на выполнение функции хеширования иногда приводят к тому, что
словарь оказывается менее производительным, чем серия операторов if. Так ли это в конкретном случае, зависит от того, какие операторы if определенной программы должны выполняться
для принятия решения. Возможно, имеет смысл выполнить тесты производительности для обоих методов в случае, если вам нужно сделать выбор между операторами if и поиском по
словарю в критически важном месте кода.
В классе CompressedGene мы широко задействовали операторы if для выбора разных вариантов методов сжатия и распаковки. Это довольно типично для Python, поскольку
в нем нет оператора switch. Вы также увидите, что для выбора из множества вариантов Python часто основательно опирается на словари вместо интенсивного использования операторов if.
Представьте себе, например, словарь, в котором мы могли бы находить соответствующую комбинацию битов для каждого нуклеотида. Иногда это выглядит более читабельным, но может снизить
производительность. Несмотря на то что с технической точки зрения сложность поиска по словарю равна O(1), затраты на выполнение функции хеширования иногда приводят к тому, что
словарь оказывается менее производительным, чем серия операторов if. Так ли это в конкретном случае, зависит от того, какие операторы if определенной программы должны выполняться
для принятия решения. Возможно, имеет смысл выполнить тесты производительности для обоих методов в случае, если вам нужно сделать выбор между операторами if и поиском по
словарю в критически важном месте кода.
На следующем шаге мы рассмотрим невскрываемое шифрование.
