На этом шаге мы рассмотрим различные способы визуализации деревьев.
Мы можем визуализировать дерево, используя функцию export_graphviz из модуля tree. Она записывает файл в формате .dot, который является форматом текстового файла, предназначенным для описания графиков. Мы можем задать цвет узлам, чтобы выделить класс, набравший большинство в каждом узле, и передать имена классов и признаков, чтобы дерево было правильно размечено:
[In 5]: from sklearn.tree import export_graphviz export_graphviz(tree, out_file="tree.dot", class_names=["nalignant", "benign"], feature_names=cancer.feature_names, impurity=False, filled=True)
[In 6]: import graphviz with open("tree.dot") as f: dot_graph = f.read() graphviz.Source(dot_graph)
Мы можем прочитать этот файл и визуализировать его, как показано на рисунке 1 используя модуль graphviz (или любую другую программу, которая может читать файлы с расширением .dot).
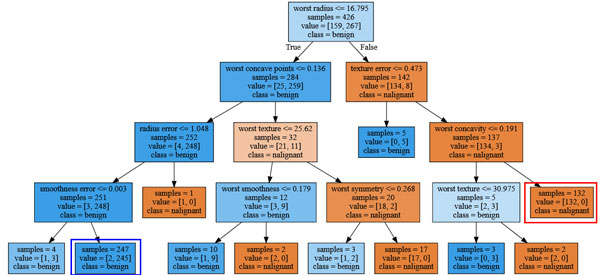
Рис.1. Визуализация дерева решений, построенного на наборе данных Breast Cancer (изображение кликабельно)
 Если вы используете Anaconda под Windows, то необходимо установить conda-пакет graphviz и pip-пакет graphviz:
Если вы используете Anaconda под Windows, то необходимо установить conda-пакет graphviz и pip-пакет graphviz:
conda install -c anaconda graphviz=2.38.0 pip install graphviz
C:\Anaconda3\Library\bin\graphviz .
Как вариант, можно построить диаграмму дерева и записать ее в файл .pdf. Дополнительно нам потребуется модуль pydotplus.
[In 7]: import numpy as np import matplotlib.pyplot as plt import pandas as pd import mglearn %matplotlib inline from sklearn.model_selection import train_test_split from sklearn.datasets import load_breast_cancer from sklearn import tree from sklearn.tree import export_graphviz cancer = load_breast_cancer() X_train, X_test, y_train, y_test = train_test_split( cancer.data, cancer.target, stratify=cancer.target, random_state=42) clf = tree.DecisionTreeClassifier(max_depth=4, random_state=0) clf = clf.fit(X_train, y_train) import pydotplus dot_data = tree.export_graphviz(clf, out_file=None) graph = pydotplus.graph_from_dot_data(dot_data) graph.write_pdf("cancer.pdf")
Можно построить визуализацию дерева с помощью функции Image интерактивной оболочки IPython:
[In 8]: from IPython.display import Image dot_data = tree.export_graphviz(clf, out_file=None, feature_names=cancer.feature_names, class_names=cancer.target_names, filled=True, rounded=True, special_characters=True) graph = pydotplus.graph_from_dot_data(dot_data) Image(graph.create_png())
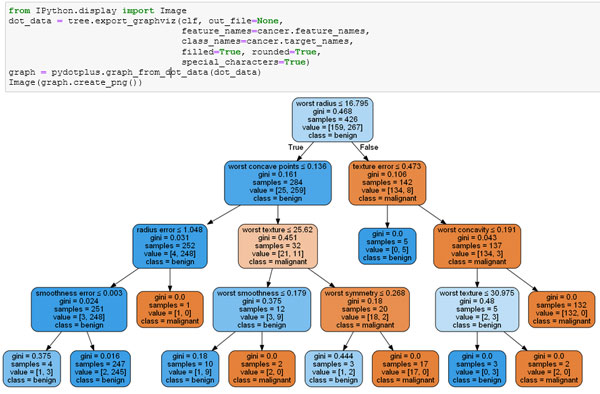
Рис.2. Визуализация дерева с помощью функции Image (изображение кликабельно)
Визуализация дерева дает более глубокое представление о том, как алгоритм делает прогнозы и является хорошим примером алгоритма машинного обучения, который легко объяснить неспециалистам. Однако, как показано здесь, даже при глубине 4 дерево может стать немного громоздким. Деревья с большим значением глубины (деревья глубиной 10 - не редкость) еще труднее понять. Один из полезных способов исследования дерева заключается в том, чтобы выяснить, какие узлы содержат наибольшее количество данных. Параметр samples, выводимый в каждом узле на рисунке 1, показывает общее количество примеров в узле, тогда как параметр value показывает количество примеров в каждом классе. Проследовав по правой ветви, отходящей от корневого узла, мы видим, что правилу worst radius>16.795 соответствует узел, который содержит 134 случая злокачественной опухоли и лишь 8 случаев доброкачественной опухоли. Далее дерево выполняет серию более точных разбиений оставшихся 142 случаев. Из 142 случаев, которые при первоначальном разбиении были записаны в правый узел, почти все (132) в конечном итоге попали в правый лист (для удобства выделен красной рамкой).
Проследовав по левой ветви, отходящей от корневого узла, мы видим, что правилу worst radius<=16.795 соответствует узел, который содержит 25 случаев злокачественной опухоли и 259 случаев доброкачественной опухоли. Почти все случаи доброкачественной опухоли попадают во второй лист справа (для удобства выделен синей рамкой), остальные случаи распределяются по нескольким листьям, содержащим очень мало наблюдений.
На следующем шаге мы рассмотрим важность признаков в деревьях.
