На этом шаге мы рассмотрим использование признаков при анализе данных.
Вместо того, чтобы просматривать все дерево, что может быть обременительно, есть некоторые полезные параметры, которые мы можем использовать как итоговые показатели работы дерева. Наиболее часто используемым показателем является важность признаков (feature importance), которая оценивает, насколько важен каждый признак с точки зрения получения решений. Это число варьирует в диапазоне от 0 до 1 для каждого признака, где 0 означает "не используется вообще", а 1 означает, что "отлично предсказывает целевую переменную". Важности признаков в сумме всегда дают 1:
[In 10]: from sklearn.model_selection import train_test_split from sklearn.datasets import load_breast_cancer from sklearn import tree from sklearn.tree import export_graphviz tree = DecisionTreeClassifier(max_depth=4, random_state=0) cancer = load_breast_cancer() X_train, X_test, y_train, y_test = train_test_split( cancer.data, cancer.target, stratify=cancer.target, random_state=42) tree.fit(X_train, y_train) print("Важности признаков:\n{}".format(tree.feature_importances_)) Важности признаков: [0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.01019737 0.04839825 0. 0. 0.0024156 0. 0. 0. 0. 0. 0.72682851 0.0458159 0. 0. 0.0141577 0. 0.018188 0.1221132 0.01188548 0. ]
Приведенная сводка не совсем удобна, поскольку мы не знаем, каким именно признакам соответствуют приведенные важности. Чтобы исправить это, воспрользуемся программным кодом, приведенным ниже:
[In 11]: for name, score in zip(cancer["feature_names"], tree.feature_importances_): print(name, score) mean radius 0.0 mean texture 0.0 mean perimeter 0.0 mean area 0.0 mean smoothness 0.0 mean compactness 0.0 mean concavity 0.0 mean concave points 0.0 mean symmetry 0.0 mean fractal dimension 0.0 radius error 0.010197368202069328 texture error 0.0483982536186494 perimeter error 0.0 area error 0.0 smoothness error 0.002415595085315826 compactness error 0.0 concavity error 0.0 concave points error 0.0 symmetry error 0.0 fractal dimension error 0.0 worst radius 0.7268285094603201 worst texture 0.045815897088866304 worst perimeter 0.0 worst area 0.0 worst smoothness 0.014157702104714051 worst compactness 0.0 worst concavity 0.0181879968644502 worst concave points 0.12211319926548449 worst symmetry 0.01188547831013032 worst fractal dimension 0.0
Мы можем визуализировать важности признаков аналогично тому, как мы визуализируем коэффициенты линейной модели (рисунок 1):
[In 12]: def plot_feature_importances_cancer(model): n_features = cancer.data.shape[1] plt.barh(range(n_features), model.feature_importances_, align='center') plt.yticks(np.arange(n_features), cancer.feature_names) plt.xlabel("Важность признака") plt.ylabel("Признак") plot_feature_importances_cancer(tree)
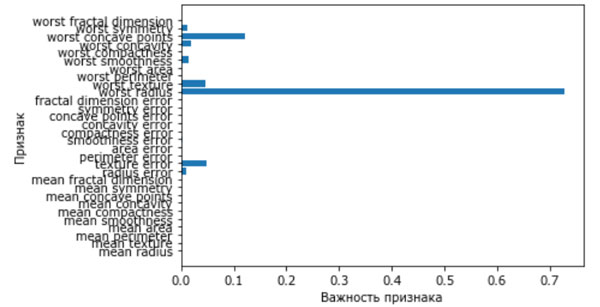
Рис.1. Важности признаков, вычисленные с помощью дерева решений для набора данных Breast Cancer
Здесь мы видим, что признак, использованный в самом верхнем разбиении ("worst radius"), на данный момент является наиболее важным. Это подтверждает наш вывод о том, что уже на первом уровне два класса достаточно хорошо разделены.
Однако, если признак имеет низкое значение feature_importance_, это не значит, что он неинформативен. Это означает только то, что данный признак не был выбран деревом, поскольку, вероятно, другой признак содержит ту же самую информацию.
В отличие от коэффициентов линейных моделей важности признаков всегда положительны и они не указывают на взаимосвязь с каким-то конкретным классом. Важности признаков говорят нам, что "worst radius" важен, но мы не знаем, является ли высокое значение радиуса признаком доброкачественной или злокачественной опухоли. На самом деле, найти такую очевидную взаимосвязь между признаками и классом невозможно, что можно проиллюстрировать на следующем примере (рисунки 2 и 3):
[In 13]: tree = mglearn.plots.plot_tree_not_monotone() display(tree)
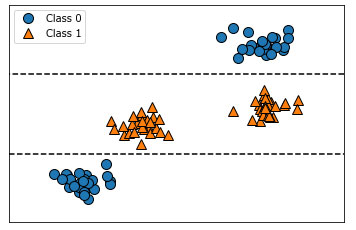
Рис.2. Двумерный массив данных, в котором признак имеет немонотонную взаимосвязь с меткой класса, и границы принятия решений, найденные с помощью дерева
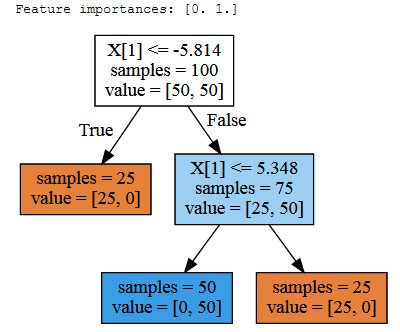
Рис.3. Дерево решений для набора данных, показанном на рисунке 2
График показывает набор данных с двумя признаками и двумя классами. Здесь вся информация содержится в X[1], а X[0] не используется вообще. Но взаимосвязь между X[1] и целевым классом не является монотонной, то есть мы не можем сказать, что "высокое значение X[0] означает класс 0, а низкое значение означает класс 1" (или наоборот).
На следующем шаге мы закончим изучение этого вопроса.
